ゲンジボタルは日本全国同じじゃない! ~ゲンジボタルに対するゲノムレベルでの集団解析を世界で初めて実施~


平素より本学の報道に関して大変お世話になっております。この度、鹿児島大学と佐賀大学、および日本ホタルの会等の研究グループが取り組んで参りました研究成果について学術論文として発表することとなりました。ぜひ、取材・報道いただきますようご案内申し上げます。
【発表概要】
鹿児島大学大学院理工学研究科(理学系)の加藤太一郎 助教らの研究グループは、佐賀大学総合分析実験センター(農学系)の永野幸生 准教授、日本ホタルの会の鈴木浩文 博士らを中心とした共同研究において、日本産ゲンジボタルのゲノムレベルでの集団解析を世界で初めて実施しました。ゲンジボタルには光り方に差、いわゆる地理的方言、があることが知られていましたが、今回の解析で日本各地に生息するゲンジボタルの細胞核DNAレベルでの遺伝的類縁関係が明らかになり、大きく3つの集団に分けられることが分かりました。またこれらの集団を簡便かつ正確に区別するDNAマーカー[1]を見出しました。ホタルの保全活動を後押しする研究成果になると期待されます。本研究成果は、英国の科学雑誌「Scientific Reports」誌(ネイチャー・リサーチ社)のオンライン版(1⽉30⽇付け)に掲載されます。
【論文情報】
雑誌名
Scientific Reports (ネイチャー・リサーチ社) (2020年1月30日オンライン掲載)
論文タイトル
Evaluation of the population structure and phylogeography of the Japanese Genji firefly, Luciola cruciata, at the nuclear DNA level using RAD-Seq analysis
著者
Dai-ichiro Kato*1, Hirobumi Suzuki2, Atsuhiro Tsuruta1, Juri Maeda1, Yoshinobu Hayashi3, Kazunari Arima1, Yuji Ito1, Yukio Nagano4 (* corresponding author)
加藤 太一郎*1、鈴木 浩文2、鶴田 篤弘1、前田 樹里1、林 良信3、有馬 一成1、伊東 祐二1、永野 幸生4 (* 責任著者)
1.鹿児島大学大学院理工学研究科(理学系)
2.日本ホタルの会
3.慶應義塾大学自然科学研究教育センター
4.佐賀大学総合分析実験センター
論文URL: https://www.nature.com/articles/s41598-020-58324-9
DOI: 10.1038/s41598-020-58324-9
報道解禁日時: 2020年1月30日19時(日本時間)
【研究の背景】
ホタルは、甲虫目ホタル科に分類される昆虫で、世界でおよそ2,800種、日本にも50種以上が生息しています。古くは日本書紀にもホタル生物発光に関する記述がみられます。その中でもゲンジボタル (学名Luciola cruciata)は日本固有種として、昔から日本人に最も親しまれてきた光る生き物の代表格です。ただし一口にゲンジボタルと言っても、日本各地に生息する本種の発光周期にはいわゆる地理的方言があることが知られていました。特に大きな違いは、新潟-長野-静岡にまたがる中部山岳地域 (フォッサマグナ地帯) を境として、発光間隔が短周期型 (2秒西日本型) と長周期型 (4秒東日本型) の2つに大別される点です。これまでの研究でこの発光周期の違いとミトコンドリアDNA[2]上のDNA配列変化(変異)には関連があることが知られていましたが、ミトコンドリアDNAよりもはるかに巨大で、より詳細な研究が可能な、細胞核DNA[2]も含めたゲノムレベルでの変異との相関関係を網羅的に調べた報告はありませんでした。
【研究成果の概要】
今回著者らは、日本各地に生息するゲンジボタルの細胞核DNA配列の違いと発光周期の地理的方言との関係性を明らかにすることを目的に、RAD-Seq(Restriction-site Associated DNA Sequencing)手法[3]を利用したゲノムレベルでの集団解析を世界で初めて実施しました(図)。本手法は、多検体のゲノムDNA配列を安価に効率よく比較できる次世代シーケンス解析方法の一つです。RAD-Seqによって得た各地のゲンジボタルの細胞核DNA配列の違い(SNPsデータ)を基に種々の解析、例えば主成分分析(PCA)や、祖先集団解析(Admixture)、遺伝的分化指標解析(Fst)、分子系統樹解析(最尤法、ベイズ法)、コアレセント解析等、を行いました。その結果、地域個体群間の遺伝的類縁関係からゲンジボタルは大きく3つの地理的集団 (東本州型、西本州型、九州型) に分けられることが明らかとなりました。
これら3つの集団のうち、西本州型と九州型の生息場所の境界はあいまいで、遺伝子の混合、つまり、互いの交雑が、境界となる山口県・佐賀県で観察されました。一方、西本州型と東本州型の生息場所はフォッサマグナ地帯を境界にはっきりと分かれており、かつ両集団間での遺伝子交雑はほとんど見られないことが
分かりました。これは、短周期型 (2秒西日本型) と長周期型 (4秒東日本型) という発光周期の異なる集団間において遺伝的交流がほぼ皆無であることを示しており、ゲンジボタル内でフォッサマグナ地帯を境とした種分化が進みつつあることがはっきりと示される結果でした。
また、これらの分子系統は、祖先型がまず東本州型と西本州-九州型に分岐し、その後西本州型と九州型に分化した、というシナリオを示唆していました。これはミトコンドリアDNAを用いたこれまでの解析結果とは異なる新たな分岐シナリオであり、ミトコンドリアゲノムと細胞核ゲノムが違った分化の過程をたどった可能性を示しています。分化シナリオと日本列島の形成過程との関連にも興味が持たれる結果でした。
一方、ゲノムレベルでの遺伝子変異を網羅的に解析したことによって、細胞核ゲノムの遺伝子配列上に3つの地理的集団を判別可能なDNAマーカーを新たに見出すこともできました。
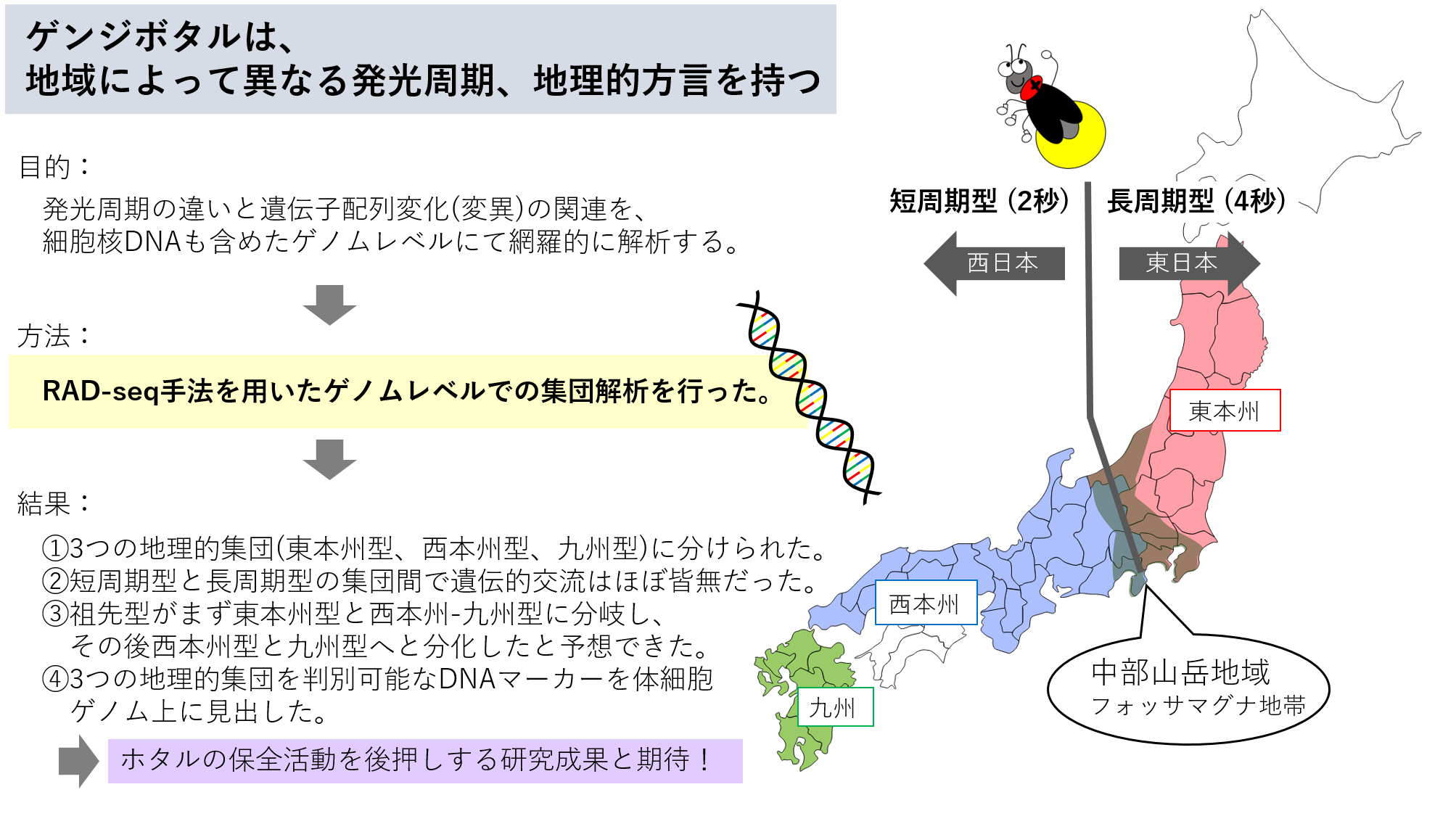
図. 本研究のまとめ
【研究の意義と展望】
本研究で、これまでのミトコンドリアDNA配列解析だけでは判定できなかった発光周期の異なるゲンジボタル間で遺伝的分化が進みつつあることを確認することができました。また、近年では自然保護や環境保全の立場から人為的な移出入による地域固有のゲンジボタルの遺伝子攪乱が危惧されていますが、その実態を正確に把握するすべはありませんでした。本研究によって得られた細胞核ゲノム上の新規DNAマーカーを利用することでこれまでよりも簡便かつ正確に3つの地理的集団を区別することが可能となるため、ホタルの保全活動を後押しする上で重要な研究成果になると期待されます。
なお、本研究は、JSPS 科研費基盤研究C (18K05320)、および公益財団法人米盛誠心育成会研究助成の支援を受けて実施されました。
【用語解説】
[1] DNAマーカー
DNAの塩基配列上の特定の位置に存在する個体の違いを示す目印(マーカー)のこと。DNAは4種類の塩基、アデニン(A)、グアニン(G)、シトシン(C)、チミン(T)、が連なってできているが、この塩基の並びが他と違っており、他と区別する際の目印となる特定の配列をDNAマーカーと呼ぶ。
[2] 細胞核DNAとミトコンドリアDNA
細胞核DNAとは真核生物の細胞核に含まれるDNAのこと。真核生物のゲノムの大部分をコードしており、両親から遺伝情報を受け継ぐ。一方、ミトコンドリアDNAとは細胞小器官であるミトコンドリア内に存在するDNAのこと。数十kb程度の環状構造で、ミトコンドリアの持つたんぱく質などに関する情報をコードしており、母方からの遺伝情報のみ受け継ぐ。これら2種類のゲノムDNA情報を利用することで、家系調査などを行うことができる。
[3] RAD-Seq(Restriction-site Associated DNA Sequencing)手法
次世代シークエンサーを利用して非常に安価に遺伝子情報を収集する方法の一つ。ゲノム上にある制限酵素サイトの周辺領域に絞ってシークエンスすることで、ゲノムの0.1~1%程度の塩基配列情報を効率的に得ることができる。領域を絞ってシークエンスするため多サンプルでの解析もコストを抑えることができ、全ゲノムを利用した解析を行うことが難しい非モデル生物に対して集団間での遺伝的変異の同定や、系統解析、集団解析等が行える。ゲンジボタルは全ゲノム情報が未公開のため、本手法を用いて遺伝子情報を収集した。
【問い合わせ先】
総合分析実験センター
准教授 永野 幸生(ながの ゆきお)
TEL: 0952-28-8898
E-mail: nagano@cc.saga-u.ac.jp